L’analyse de la robustesse par «bootstrap»
Pour compléter la construction de l'arbre phylogénétique avec les méthodes énoncées ci-dessus, une analyse de la robustesse doit être effectuée, d'habitude par le « bootstrap ». Cette technique est un dérivé des simulations de Monte-Carlo, qui consiste à échantillonner les positions de l'alignement pour relancer la construction phylogénétique de façon itérative puis de comparer les résultats obtenus après plusieurs répétitions (n >1000) Le résultat est représenté sous la forme d'un arbre consensus dans lequel figurent les regroupements majoritairement apparus. Une valeur de « bootstrap » (pourcentage de 0 à 100%) est associée à chaque branche de l'arbre indiquant le nombre de fois où cette branche a été retrouvée au fil des répétitions et juger ainsi de leur crédibilité. En d'autres termes, la valeur de « bootstrap » indique une évaluation de la résistance d'un noeud à la perturbation des données.
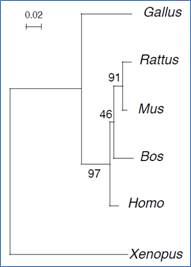
Figure 7: Arbre « bootstrappé » (Pierre-Henri Gouyon)
Sur certains arbres, la valeur « bootstrap » au-dessus indique que le support est par rapport aux exons et introns en même temps, alors que la valeur au-dessous indique que le support est seulement pour les exons.
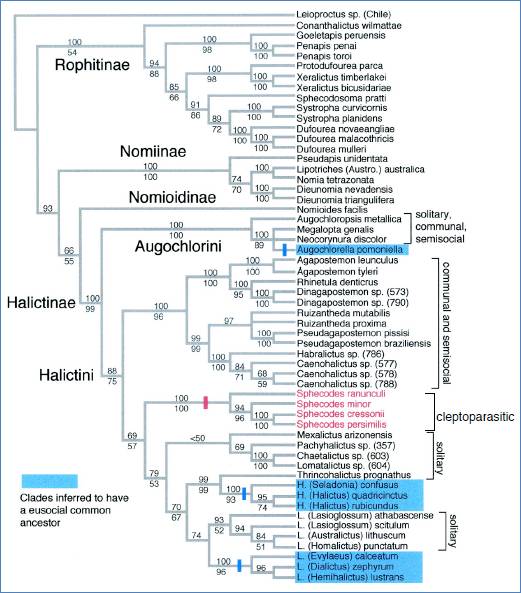
Figure 8: Arbre phylogénétique (Danforth, r12)