Galaxy
Galaxy est développé conjointement par le Nekrutenko Lab (Center for Comparative Genomics and Bioinformatics , Penn State University) et le Taylor Lab (Emory University). Il s'agit d'un logiciel entièrement libre et gratuit, écrit en Python et destiné à faciliter la manipulation et l'analyse des données. Elle permet d'utiliser des logiciels habituellement exécutés en ligne de commande de manière graphique, grâce à un système de plugins en XML et de gabarits Mako. Ces outils sont particulièrement puissants et par défaut sont massivement adaptés au traitement et à l'analyse des données génomiques (SAMtools , ClustalW , EMBOSS , Bowtie , etc.)
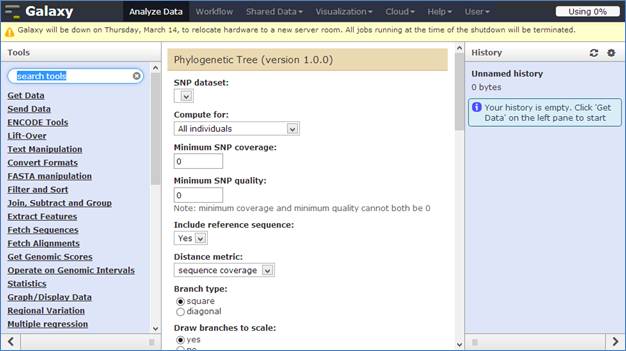
Figure 12: Interface principale du logiciel Galaxy
Galaxy peut être installé dans l'infrastructure locale, ou utilisé à travers deux instances publiques accessibles à tous, entretenues par l'équipe de Galaxy. Les instances publiques permettent aux visiteurs d'effectuer des analyses gratuitement dans la mesure de la disponibilité des ressources disponibles sur les serveurs de calcul, ou encore permet d'utiliser des ressources distantes pour le calcul, telles que celles proposées par Amazon Elastic Compute Cloud avec la version Galaxy Cloudman, mais plus dispendieuses.
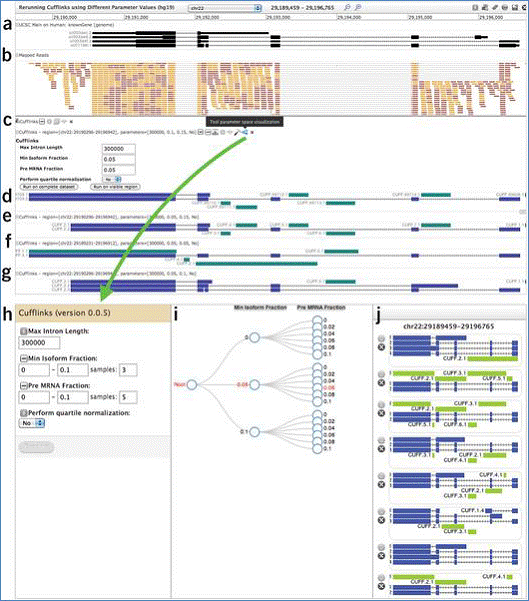
Figure 13: Exemple de sous-flux de travaux
(Goecks et al., r18)
de la présentation d'Étienne Lord (Étienne Lord, r3)